

Actualizado el miércoles, 15 febrero 2006
2.2 RECOMBINACIÓN GENERAL U HOMÓLOGA
2.3 RECOMBINACIÓN ESPECÍFICA DE SITIO CONSERVATIVA
2.3.1 INTEGRACIÓN DEL GENOMIO DEL FAGO l EN EL CROMOSOMA DE E. COLI
2.3.2 INVERSIÓN DE SEGMENTOS DE ADN POR RECOMBINACIÓN ESPECÍFICA CONSERVATIVA
2.4 RECOMBINACIÓN ESPECÍFICA “ILEGÍTIMA”: PROPICIADA POR ELEMENTOS GENÉTICOS TRANSPONIBLES
2.4.2 TIPOS DE ELEMENTOS GENÉTICOS TRANSPONIBLES EN BACTERIAS
2.4.3 CARACTERISTICAS GENERALES DE LA TRANSPOSICION
2.4.4 REGULACION DE LA TRANSPOSICION Y POSIBLE SIGNIFICADO EVOLUTIVO
3 SISTEMAS DE RESTRICCIÓN Y MODIFICACIÓN
4.1 INTRODUCCIÓN A LA TRANSFORMACIÓN (CONCEPTO Y APROXIMACIÓN HISTÓRICA)
4.2.1 CARACTERES GENERALES DE LA TRANSFORMACIÓN NATURAL
4.2.2 TRANSFORMACIÓN EN BACTERIAS GRAM POSITIVAS
4.2.2.1 EN STREPTOCOCCUS PNEUMONIAE
4.2.2.1.1 DESARROLLO DE LA COMPETENCIA (Y MODO DE UNIÓN DEL ADN A LA SUPERFICIE CELULAR)
4.2.3 TRANSFORMACIÓN EN BACTERIAS GRAM NEGATIVAS
4.2.3.1 TRANSFORMACIÓN EN HAEMOPHILUS Y NEISSERIA
![]()
En la introducción al capítulo 15 ya realizamos un encuadre general de los distintos tipos de variaciones genotípicas, distinguiendo las que no implican transferencia de material genético (mutación y curación) de las que derivan de transferencia de ADN entre bacterias. A partir de ahora nos vamos a ocupar de estas últimas.
Rasgos generales de la transferencia genética en bacterias:
|
La transferencia es unidireccional, es decir, tiene una determinada polaridad, existiendo células donadoras y células receptoras. | |
|
La transferencia del genomio de una célula a otra no suele ser total, sino parcial. | |
|
Parte del material genético, una vez introducido en la célula receptora, sufre inmediatamente un fenómeno de recombinación con el genomio de la receptora. El resto del material de la donadora o no se replica, o se ve destruido. | |
|
Como se puede deducir, tras los procesos de transferencia genética bacteriana, no surge una diploidía total (como es el caso en eucariotas), sino una diploidía parcial, que recibe el nombre de merodiploidía. Las células bacterianas diploides parciales reciben la denominación de merodiploides o merozigotos. Además, la merodiploidía suele ser transitoria. |
Este tipo de intercambio genético unidireccional, con diploidía transitoria parcial, se denomina meromixia, para distinguirlo de la reproducción sexual de eucariotas.
|
El genomio de la célula receptora se suele denominar endogenote. | |
|
La porción de genomio de la cél. donadora que se transfiere se llama exogenote. |
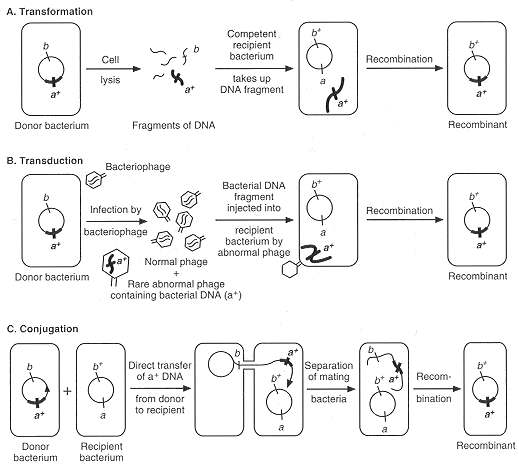
Los procesos de transferencia genética en bacterias son de tres grandes tipos, más una variante adicional de uno de ellos:
|
TRANSFORMACIÓN: captación y asimilación de ADN libre (desnudo), a partir del medio, por parte de una célula receptora (lo estudiaremos en la segunda parte de este capítulo). | |
|
CONJUGACIÓN: transferencia directa de material genético, promovida por un plásmido, desde una célula donadora a otra receptora, por medio de contactos íntimos entre ambas (puentes de unión). Una variante de la conjugación es la SEXDUCCIÓN: en ella, un trozo definido de mat. genético de la donadora es transferido como parte de un plásmido conjugativo (cap. 18). | |
|
TRANSDUCCIÓN: el material genético es transportado desde la cél. donadora a la receptora por medio de un virus bacteriano (o sea, un bacteriófago o simplemente, fago), que actúa como vector (ver cap. 19). |
Pero antes de abordar estos sistemas de transferencia genética, vamos a considerar el destino posible del material genético transferido por estos procesos (primera parte del presente tema). Dejando aparte la posibilidad de que el material transferido sea por sí mismo un replicón, y por lo tanto, al llegar a la célula receptora pueda replicarse y mantenerse por sí mismo, el exogenote puede sufrir dos destinos muy distintos:
|
Por un lado, puede verse implicado en un tipo de proceso de recombinación genética, que lo “incorpora” de alguna forma al endogenote. | |
|
Por otro lado, el exogenote puede ser “reconocido como extraño” por parte de la célula receptora, en cuyo caso será destruido. |
La recombinación es el proceso por el que la información genética se ve redistribuida, tras la transferencia del exogenote al endogenote. La recombinación permite “barajar” grandes conjuntos de genes, suministrando una fuente de variación y selección para la evolución, más rápida que la mutación.
En bacterias encontramos los siguientes tipos principales de recombinación:
1) Recombinación general u homóloga
2) Recombinación específica (no-homóloga), en la que a su vez distinguimos:
a) Rec. específica legítima, conservativa;
b) Rec. específica “ilegítima”, propiciada por elementos genéticos transponibles.
A continuación, y antes de abordar cada tipo de recombinación, daremos las definiciones generales de cada uno de estos procesos recombinativos:
Recombinación general u homóloga
|
Puede ocurrir en cualquier lugar del genomio, por emparejamiento entre pares de secuencias que presenten una homología suficientemente extensa. | |
|
Depende de la intervención de un equipo enzimático característico, en el que la proteína RecA (o equivalente) es central en el proceso. |
Recombinación específica legítima (conservativa)
|
Requiere cortas secuencias de homología entre el exogenote y el endogenote (por eso se llama también “específica de sitio”), que son reconocidas por proteínas específicas. | |
|
Es independiente de RecA. | |
|
Este tipo de recombinación es típica de la integración de genomios de fagos en sitios concretos del genomio de bacterias hospedadoras. |
Recombinación específica ilegítima: fenómenos de transposición
|
No depende de homologías (ni siquiera cortas) entre el exogenote (en este caso, un elemento genético transponible, como IS o Tn) y el endogenote. | |
|
También es independiente de RecA. | |
|
El elemento transponible codifica una enzima (genéricamente se les denomina transposasas) que reconoce secuencias específicas inversamente repetidas en los extremos del propio elemento, lo cual se requiere para el proceso de transposición. | |
|
Muchos elementos transponibles (pero no todos) poseen un mecanismo replicativo de transposición: es decir, al transponerse dejan una copia de sí mismos en el sitio original, y generan una copia nueva en el sitio a donde se transponen (recombinación específica duplicativa). |
Anteriormente ya definimos sus características generales. A continuación vamos a describir un modelo molecular de esta recombinación, basado en el clásico de Meselson y Radding, modificado con los últimos avances. No se olvide que estamos ante un modelo, es decir, una propuesta hipotética para explicar un conjunto de datos experimentales. No todos los puntos de este modelo están totalmente aclarados o demostrados:
Supongamos que tenemos un exogenote y un endogenote, ambos consistentes en dobles hélices. En los modelos de recombinación, al exogenote se le suele denominar como ADN donador, y al endogenote como ADN receptor.
1) Inicio de la recombinación: La recombinación homóloga comienza con una incisión endonucleotídica en una de las cadenas de la doble hélice donadora. La responsable de este proceso es la nucleasa RecBCD (=nucleasa V), que actúa de la siguiente manera: se une aleatoriamente al ADN del donador, y se va moviendo por la doble hélice hasta que encuentra una secuencia característica denominada c (letra griega “chi”), que es un “punto caliente recombinativo”([1]):
5' GCTGGTGG 3'
3' CGACCACC 5'
Una vez reconocida la secuencia, la nucleasa RecBCD corta a 4-6 bases a la derecha (lado 3') de la cadena superior (tal como las hemos escrito arriba). Entonces, esta misma proteína, actuando ahora como una helicasa, va desenrollando la cadena cortada, haciendo que se desplace una zona de ADN de cadena sencilla (ADN c.s.) con su extremo 3’ libre
2) El hueco que ha dejado la porción desplazada de la cadena cortada del donador es rellenado mediante síntesis reparativa de ADN.
3) La zona de cadena sencilla desplazada del ADN donador es recubierta por subunidades de la proteína RecA (a razón de un monómero de RecA por cada 5-10 bases). De este modo, esa cadena sencilla adopta una configuración helicoidal extendida.
4) Asimilación o sinapsis: Este es el momento clave de actuación de la RecA. De alguna manera, la RecA unida al ADN c.s. del donador facilita el encuentro de éste con la parte de doble hélice complementaria del receptor, de modo que en principio se forma una triple hélice. A continuación, con la hidrólisis de ATP, la RecA facilita que la cadena del donador desplace a la cadena homóloga del receptor, y por lo tanto se empareje con la complementaria de ese receptor. En este proceso, la porción de cadena del receptor homóloga del donador se ve desplazada, originándose la llamada “estructura en D”.
|
Es importante resaltar que este proceso propiciado por RecA depende de que el donador y el receptor presenten gran homología de secuencia (del 100 al 95%), y de que estos segmentos de homología tengan más de 100 bases de longitud. | |
|
Observar que esta sinapsis implica la formación de una porción de heterodúplex en la doble hélice receptora: hay una zona en la que cada cadena procede de un ADN c.d. parental distinto (donador y receptor). |
5) Se supone que la cadena recién desplazada del ADN receptor (estructura en D) es digerida por nucleasas.
6) Unión covalente de los extremos originados en las dos cadenas homólogas. Esto da como resultado un entrecruzamiento simple por el que las dos dobles hélices se encuentran “atadas”. La estructura global resultante se denomina estructura o juntura de Holliday.
7) Migración de las ramas: al punto de cruce de la estructura de Holliday se une un complejo formado por las proteínas RuvA y RuvB, que con hidrólisis de ATP logran el desplazamiento del punto de cruce de Hollyday: de esta forma se va agrandando la porción de heterodúplex en ambas dobles hélices.
8) Isomerización: para visualizarla fácilmente, imaginar que rotamos los dos segmentos de uno de los ADN c.d. 180o respecto del punto de entrecruzamiento, para generar una estructura plana que es isómera de la anterior (“estructura en X”).
9) Resolución de esta estructura: este paso está catalizado por la proteína RuvC, que corta y empalma entre sí dos de las cadenas entrecruzadas en la juntura de Hollyday. El resultado de la resolución puede variar según que se corten y empalmen las cadenas que antes no participaron en el entrecruzamiento, o que vuelvan a ser éstas las implicadas en esta segunda operación de corte y sellado:
a) Si los cortes y empalmes afectan a las cadenas de ADN que antes no participaron en en el entrecruzamiento, el resultado será dos moléculas recombinantes recíprocas, donde cada una de las 4 cadenas son recombinantes (ha habido intercambio de marcadores entre donador y receptor)
b) Si los cortes y empalmes afectan a las mismas cadenas que ya habían participado en el primer entrecruzamiento, el resultado consistirá en dos dobles hélices que presentan solamente sendas porciones de ADN heterodúplex.
Este modelo, como dijimos es aplicable a bacterias. Sin embargo, dejando aparte los aspectos enzimáticos (proteínas Rec y Ruv), los aspectos mecánicos (principalmente los requerimientos de grandes zonas de homología, sinapsis, estructuras de Holliday, formación de heterodúpex, etc.) son aplicables también a los organismos superiores (quizá el alumno haya estudiado o estudiará un modelo parecido en la asignatura de Genética). De todas formas, hay que volver a insistir en una notable diferencia entre la recombinación homóloga de las bacterias y la de los organismos superiores de reproducción sexual: en las bacterias la recombinación afecta a porciones relativamente pequeñas del genoma donador, mientras que en los eucariotas cada cromosoma completo se empareja con el homólogo (si bien sólo se producen unos pocos entrecruzamientos).
Caracteres generales:
|
Es independiente de RecA (u otras enzimas de rec. homóloga). | |
|
Es independiente de grandes regiones de homología entre exo- y endogenote. | |
|
El proceso está catalizado por enzimas específicas llamadas en general “recombinasas”, que tienen un mecanismo de acción similar al de las topoisomerasas de tipo I. | |
|
No hay replicación de ADN durante el evento de recombinación específica de sitio, por lo que también se la llama “conservativa”. |
Estudiaremos dos ejemplos de esta clase de recombinación: el clásico caso de la integración del genomio del fago l en un punto concreto del cromosoma de E. coli, y la variación de fase flagelar por inversión de un segmento de ADN de Salmonella typhimurium.
Existen otros elementos genéticos que codifican integrasas: los denominados integrones y algunas islas genómicas (ver capítulo 7).
Introducción al concepto de fago moderado (avance de lo que veremos en la sección de Virología):
|
Ciclo lítico (vegetativo) y ciclo lisogénico. | |
|
En el estado lisogénico, el ADN de l se integra en el cromosoma de E. coli, y mantiene todas sus funciones líticas “desconectadas”. En esta situación se le llama profago, y se comporta como una porción “normal” del genomio de su hopedador. Eventualmente, el profago puede inducirse, es decir, sus funciones líticas se activan: el profago se escinde del cromosoma bacteriano, se circulariza y conecta todos los genes cuya expresión conducirá a la producción de muchas partículas del virus, que terminan lisando la célula, y liberándose para reiniciar el proceso. | |
|
Observen las diferencias en el mapa del ADN del fago: cuando se encuentra dentro de la partícula del virus (ADN c.d. lineal: A.....attP....R), pero al entrar a la bacteria, se circulariza. Como profago también es lineal, pero con un orden distinto al de la partícula del fago antes de la infección (......A R........). |
El paso del ADN del fago desde su forma circular (vegetativa) a la forma de profago (ciclo lisogénico) se debe a un fenómeno de recombinación específica de sitio, en el que están implicadas las secuencias attP (del fago) y attB (de la bacteria). La zona attB está situada entre los operones gal y bio del cromosoma de E. coli.
Cada una de las zonas att está compuesta de una secuencia central (núcleo, “O”), de 15 p.b., y de dos regiones adyacentes, llamadas brazos. Los núcleos de attP y de attB son exactamente idénticos, mientras que los 4 brazos (los dos del fago, P y P', y los dos de la bacteria, B y B') son diferentes del núcleo y diferentes entre sí.
El evento de integración se puede representar así:
attP X attB à attL + attR
Si atendemos a la nomenclatura a base de núcleos y brazos, tenemos:
POP' X BOB' à BOP' + POB'
Este proceso requiere la actuación de dos tipos de proteínas:
|
Int: la integrasa, codificada por el gen int del fago l. | |
|
IHF (=factor de integración del hospedador), codificado por dos genes de la bacteria (himA, himD). |
La escisión es, desde el punto de vista “mecánico”, la inversa de la reacción anterior para regenerar el ADN circular del fago, mediante la recombinación específica:
attL X attR à attP + attB
(BOP' X POB' à POP' + BOB')
Ahora bien, para esta reacción hace falta, aparte de las proteínas Int e IHF, una segunda proteína producida por l : proteína Xis (llamada también escisionasa). La razón de una proteína adicional para revertir el proceso de la integración cobra su sentido si pensamos en que la combinación de brazos que rodea a los núcleos es distinta en la integración y en la escisión. La proteína Xis confiere la “especificidad” para reconocer las secuencias attL y attR. Por lo demás el proceso de escisión implica un mecanismo similar al de la integración (que es lo que vamos a estudiar a continuación).
Aspectos moleculares de la integración del fago l
Se reconocen cortas secuencias de homología entre el ADN del fago y de la bacteria: concretamente las zonas homólogas “O” tienen solamente 15 p.b.
Se producen cortes específicos en cada una de las cadenas, en posiciones equivalentes, de modo que quedan extremos protuberantes 5' con 7 nucleótidos en cadena sencilla.
La reacción de la integrasa se parece a la de las topoisomerasas de tipo I: rompe una cadena de ADN en cada uno de los sitios att, manteniendo fijos los extremos recién cortados. La cadena intacta de cada sitio att pasa a través del hueco abierto. A continuación, el extremo 3' de un sitio att es unido al extremo 5' del otro sitio att. La operación que acabamos de describir se repite con las otras cadenas que antes habían quedado intactas. Al igual que las topoisomerasas de tipo I, la integrasa no requiere ATP, ya que la energía del enlace fosfodiéster cortado se conserva en forma de enlace covalente entre el extremo recién cortado y la propia enzima. Esta “energía” es la que luego se emplea en la operación de empalme (creación de un nuevo enlace fosfodiéster).
El proceso que hemos descrito ocurre por medio de una gran partícula formada por muchas unidades de Int y de IHF (unas 70 copias de IHF y de 20-40 de Int). Esta partícula reconoce primero el sitio attP (del fago), siempre que el ADN esté superenrollado negativamente. El ADN del fago de la zona attP se arrolla alrededor de la partícula multiproteica, a la manera en la que se empaqueta el ADN en los nucleosomas. Por esta razón, a esta partícula que lleva arrollado el ADN del fago se la denomina intasoma. Una vez que el attP ha establecido los contactos específicos con las proteínas Int e IHF del intasoma, éste “captura” a la secuencia attB de la bacteria, haciendo que ambas att queden perfectamente alineadas para proceder a la recombinación específica.
(Nota aclaratoria: lo que vamos a describir a continuación es un proceso de recombinación específica dentro de un mismo genomio. O sea, aquí no podemos hablar estrictamente de exogenote ni endogenote, ya que no hay un fenómeno previo de transferencia de ADN. Sin embargo, lo estudiamos en esta sección, ya que el mecanismo es del mismo tipo que el que acabamos de ver para la integración del fago l). Además esta inversión sirve como mecanismo de adaptación al medio, ya que permite la regulación de ciertos genes bajo determinadas condiciones.)
Desde hace tiempo se venía observando que ciertas propiedades de algunas bacterias cambian su fenotipo con una frecuencia varios órdenes de magnitud superior a la tasa de mutación espontánea. Además, se da la circunstancia de que estos cambios son también reversibles. Algunos ejemplos:
|
antigenicidad flagelar; | |
|
antigenicidad de pelos (fimbrias adhesivas); | |
|
adhesividad; | |
|
virulencia de ciertas cepas patógenas, etc. |
Se ha visto que muchos de estos fenómenos de inestabilidad fenotípica reversible se deben a un mecanismo de recombinación específica de sitio, por el que un segmento definido de ADN cambia su orientación (o sea, se invierte respecto de su polaridad anterior). Uno de los casos mejor estudiados es el de la transición de fase flagelar en Salmonella typhimurium:
Dado un clon original homogéneo respecto del tipo de flagelina, al cabo de poco tiempo de crecimiento, se puede observar que se han producido dos subpoblaciones diferenciadas respecto del tipo de flagelina:
|
subpoblación con flagelos a base de flagelina H1; | |
|
subpoblación con flagelos a base de flagelina H2. |
Cada flagelina se produce por un gen distinto.
La transición desde un tipo de flagelina al otro se da con alta frecuencia (aprox. 10--3, frente a sólo 10--7 para la frecuencia de mutación espontánea). Veamos cómo ocurre:
El segmento invertible contiene el gen hin (que deriva su nombre de H-inversion), el cual codifica una recombinasa específica (“invertasa”), que reconoce los terminales inversamente repetidos (IR) de 14 pb que limitan a dicho segmento. El efecto de la enzima es aproximar entre sí a los dos IR, para luego efectuar los cortes y empalmes específicos, actuando como una topoisomerasa de tipo I.
Otros ejemplos de inversión específica de segmentos:
|
En ciertos fagos existe un segmento invertible de ADN, de modo que en cierta orientación el fago infecta a determinadas especies y cepas bacterianas, mientras que la otra orientación le suministra al fago la capacidad de infectar otras cepas e incluso especies distintas (ello se debe a que en cada orientación el segmento induce la síntesis de tipos distintos de fibras para adsorberse sobre distintas bacterias) . Por ejemplo, el fago Mu: posee el segmento “G”, invertible por el producto del gen gin; ello le permite cambiar de bacteria hospedadora (puede cambiar de Escherichia coli a Citrobacter); el fago P1: posee el segmento “C”, invertible por el producto del gen cin. Los genes hin, gin, cin y otros están relacionados evolutivamente entre sí y con otros genes codificadores de recombinasas específicas, incluyendo las resolvasas de algunos transposones (las resolvasas serán estudiadas en el apartado sobre los elementos genéticos transponibles). | |
|
La síntesis de fimbrias adhesivas de ciertas cepas patógenas de Escherichia coli viene regulada por una secuencia invertible. |
Significado evolutivo del mecanismo de inversión específica de ADN:
|
Permite cambios adaptativos rápidos y reversibles ante posibles cambios rápidos en determinadas condiciones del medio ambiente que son importantes dentro del nicho ecológico de la bacteria. | |
|
Algunas bacterias patógenas usan este sistema para escapar al sistema inmunitario del hospedador al que parasitan. Por ejemplo, la variación de fase de fimbrias en Neisseria: en algunas especies patógenas de este género (gonoco, p. ej.) existen varios genes de pilina, cada uno capaz de codificar una pilina antigénicamente distinta de las demás. Cada clon de la bacteria transcribe en cada momento solamente uno de estos genes, permaneciendo los demás silenciosos. Pero con una elevada frecuencia (10--3) se producen recombinaciones específicas entre una copia silenciosa y la versión que hasta el momento estaba activa. Esto hace que a partir de ahora sea la nueva versión la que se exprese: da origen a una nueva pilina, para la que el sistema inmune del hospedador no tiene anticuerpos, lo que da una ventaja a la bacteria en su capacidad de patogenia. |
Los elementos genéticos transponibles “saltan” de un lugar a otro de los genomas por un mecanismo de recombinación no homóloga “ilegítima”, es decir, que no depende de homologías entre el elemento y la zona de genomio a la que este elemento va a transponerse (esta última se suele denominar secuencia diana).
En general, las enzimas que catalizan ese proceso se denominan transposasas. La transposasa corta el ADN “donador” en los extremos del elemento transponible, y luego los inserta en el ADN diana, aunque los detalles varían entre las distintas clases de elementos móviles. La mayor parte de los elementos transponibles permanecen unidos durante el evento de transposición a ADN contiguo (es decir, nunca llegar a estar “libres”, sino permanecen ligados por alguna de sus cadenas al ADN donador o al ADN diana).
Muchos elementos transponibles bacterianos tienen un mecanismo replicativo: la transposición se realiza por un tipo especial de replicación del elemento transponible, de modo que queda una copia del mismo en la posición original, mientras otra copia se inserta en una nueva posición, bien sea del mismo replicón, bien sea de otro replicón distinto del original y presente en la misma célula (p. ej., un plásmido). Por ello se habla en estos casos de recombinación ilegítima duplicativa. Pero también hay elementos transponibles que siguen un modelo no duplicativo, a veces llamado de “cortar y pegar”, porque el elemento se escinde de su localización original y se inserta en una nueva.
Todos los elementos genéticos transponibles poseen terminales inversamente repetidos (IR), es decir, la secuencia de un extremo en una cadena, leída en sentido 5’à3’ es idéntica (o casi) a la secuencia del otro extremo de la otra cadena, también leída en su sentido 5’à3’.
Un rasgo general de la transposición es que una vez acabada ésta, el elemento móvil aparece “emparedado” entre copias directas de la secuencia diana. Ello se debe a que la transposición implica la duplicación de dicha secuencia. Las secuencias diana suelen ser de pocos pares de bases, y aunque dicha secuencia no suele ser específica (varía de un evento a otro de transposición del mismo elemento), cada elemento transponible suele conllevar la duplicación de secuencias de una longitud determinada (algunos elementos tienen dianas de 4 pb., mientras otros las tienen de 9 pb, etc.).
Aunque el lugar de la transposición no tiene especificidad de secuencia diana, tampoco es un fenómeno aleatorio. Se sabe que algunos transposones prefieren insertarse en sitios con determinadas propiedades de ADN, como grado de superenrollamiento, de metalición, o susceptibilidad a la transcripción.
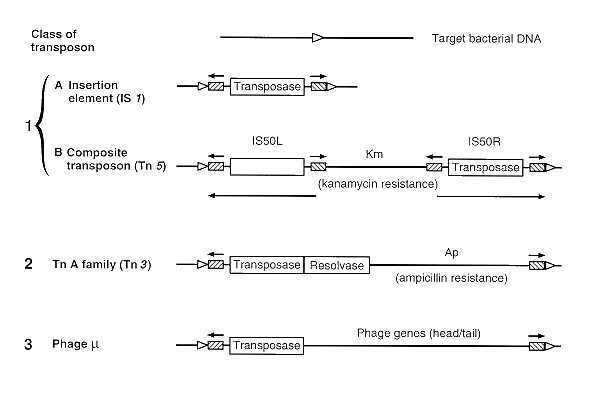
Secuencias de inserción (IS):
|
Son elementos pequeños (del orden de 1000 pb., o menos), dotadas en sus extremos de terminales inversamente repetidos (IR), de unos 15 a 25 pb. | |
|
Normalmente sólo poseen un gen, que codifica la transposasa. Por lo tanto, a diferencia de los transposones, no confieren fenotipo seleccionable. | |
|
En los genomas de muchas bacterias existen varias copias de cada tipo de IS. Por ejemplo, en Escherichia coli K12 existen unas 6 copias de IS1, 7 de IS2, etc. Los plásmidos, especialmente los conjugativos suelen ser ricos en IS (un ejemplo de esto lo veremos en el tema 18, cuando hablemos del plásmido F de E. coli). | |
|
La frecuencia de transposición varía según el tipo de IS, pero en general oscila entre 10--3 y 10--4 por división celular. La inserción de una IS en un gen concreto ocurre a una frecuencia de 10--5 y 10--7, es decir, del orden de la frecuencia de mutaciones espontáneas puntuales. La reversión de la inserción (por escisión exacta de la IS) es menos frecuente: 10--6 a 10--10 por generación. |
Transposones (Tn):
Son elementos de mayor tamaño que los IS (desde casi 3.000 pb hasta más de 20.000 pb). Poseen al menos un gen para la transposasa y un gen responsable de alguna función no relacionada con la transposición: concretamente, muchos transposones portan genes cuya expresión da fenotipos fácilmente seleccionables o detectables (p. ej.: resistencia a antibióticos o a metales pesados). Estos genes marcadores son muy útiles, ya que facilitan grandemente el estudio de estos transposones en laboratorio.
Dentro de los transposones bacterianos podemos distinguir dos subtipos principales:
|
Transposones compuestos, cuyos extremos son dos secuencias de inserción idénticas o casi idénticas. Dentro de esta clase, algunos poseen las dos copias de IS en la misma orientación, mientras que otros las poseen en orientaciones opuestas; sin embargo, como a su vez las IS poseen cortos terminales inversamente repetidos (IR), todos los transposones citados tienen este tipo de secuencias IR. Se comportan como las respectivas IS, que son las responsables de la transposición. Precisamente, la transposición depende de una o de ambas copias de la IS. En su mecanismo de transposición pueden dar, como productos finales, cointegrados de replicones y transposiciones. Ejemplos:
| |||||||
|
Transposones no compuestos: carecen de IS, y sus extremos son dos cortas secuencias inversamente repetidas (IR). Llevan en su porción central un gen para la transposasa, y a veces también un gen para la resolvasa y/o para la regulación de la transposición. Suelen dar sólo transposiciones, siendo los cointegrados únicamente una fase transitoria que actúan como intermediarios del mecanismo de transposición. Los cointegrados se deshacen (se “resuelven”) por una resolvasa codificada por el mismo transposón. Existen muchos ejemplos de este tipo, que se pueden agrupar en “familias” que presentan un origen evolutivo común: Citaremos la denominada familia de TnA, que incluye Tn1 (AmpR), Tn3 (AmpR), Tn1000, etc. Los miembros de una misma familia comparten el mismo tipo de sistema de transposa/resolvasa, pero cada miembro puede contener genes de resistencia (u otros) totalmente diferentes, que han debido de aquirir independientemente a lo largo de la evolución. Una clave de cómo han podido adquirirlos la da una categoría especial de transposones, llamada integrones. |
Integrones (In):
![]() Los integrones son un nuevo tipo de elementos transponibles dotados de
terminales inversamente repetidos (y a menudo con marcadores de resistencia a
antibióticos o a metales pesados) que se caracterizan por poseer un gen
codificador de una integrasa. Se transponen como una IS o un Tn, pero además,
la integrasa les ha capacitado evolutivamente para realizar una recombinación
específica de sitio (al estilo de la Int del fago l) por la que han incorporado a su
estructura algún gen procedente de otro elemento genético. Los integrones
pueden a su vez formar parte de transposones mayores (p. ej., el Tn21 es un gran transposón que codifica múltiples resistencias a
antibióticos, y dentro de él se encuentra el integrón In2, que contiene su propio gen int
(integrasa), que se sitúa al lado de un gen de resistencia a estreptomicina
“capturado” (por recombinación específica) de otro elemento genético (se
ignora de dónde).
Los integrones son un nuevo tipo de elementos transponibles dotados de
terminales inversamente repetidos (y a menudo con marcadores de resistencia a
antibióticos o a metales pesados) que se caracterizan por poseer un gen
codificador de una integrasa. Se transponen como una IS o un Tn, pero además,
la integrasa les ha capacitado evolutivamente para realizar una recombinación
específica de sitio (al estilo de la Int del fago l) por la que han incorporado a su
estructura algún gen procedente de otro elemento genético. Los integrones
pueden a su vez formar parte de transposones mayores (p. ej., el Tn21 es un gran transposón que codifica múltiples resistencias a
antibióticos, y dentro de él se encuentra el integrón In2, que contiene su propio gen int
(integrasa), que se sitúa al lado de un gen de resistencia a estreptomicina
“capturado” (por recombinación específica) de otro elemento genético (se
ignora de dónde).
|
Las transposasas reconocen los terminales inversamente repetidos del transposón, y cortan en los extremos (en este sentido se dice que la transposición es específica, pero sólo respecto de las secuencias del elemento transponible). | |
|
Por otro lado, las transposasas cortan también en una secuencia diana más o menos aleatoria del sitio a donde se van a transponer (inespecificidad respecto del “endogenote”, por lo que se llama a esta recombinación “ilegítima”). Los cortes son en ambas cadenas, pero en situaciones ligeramente separadas entre sí en las dos cadenas. Posteriormente hay empalmes entre las cadenas cortadas del Tn y de la diana. | |
|
La transposición duplicativa implica la replicación de la corta secuencia diana (dependiendo del transposón puede ser de 4 hasta 12 pb.) y la replicación del propio transposón (por ello se habla de recombinación ilegítima duplicativa). | |
|
Al final de una transposición duplicativa encontramos una copia del Tn en el sitio original, y otra copia en el sitio receptor, limitada por sendas repeticiones directas de la corta secuencia diana (como acabamos de decir, una de estas repeticiones surge por replicación durante el proceso de la transposición). |
Veamos un poco más en detalle el mecanismo de transposición de un transposón típico, el Tn3
Breve descripción del Tn3: es un transposón de una 5 kb., cuyos extremos son terminales inversamente repetidos de 38pb. Posee 3 genes, de los cuales dos están implicados en la transposición:
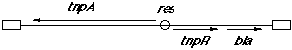
|
tnpA: codifica la transposasa (enzima de recombinac. “ilegítima”, propia de este Tn). | |
|
tnpR: codifica la resolvasa (enzima de tipo recombinasa específica de sitio-conservativa, parecida a las que hemos estudiado en el apartado 2.3). Como veremos, la resolvasa actúa también como reguladora de la transposición). | |
|
bla: codifica una b-lactamasa (responsable del rasgo fenotípico AmpR de resistencia a la ampicilina conferido por este transposón). | |
|
Observar que, además, existe una secuencia no codificadora, llamada res (=IRS), que como veremos, interviene durante la fase de resolución de los cointegrados. |
Modelo de transposición de Tn3: ocurre en dos etapas:
1) Formación de un cointegrado entre el replicón donador y el replicón receptor, reacción que depende de la transposasa. En más detalle:
a) la transposasa reconoce los terminales IR del Tn3, y realiza dos cortes, uno en cada cadena, pero cada corte en un extremo distinto;
b) la transposasa corta en el ADN diana, en cada cadena, en posiciones separadas por unos pocos nucleótidos;
c) cada extremo protuberante de la secuencia diana se une a un terminal cortado del Tn3. Observar que en este momento tenemos una estructura en X, pero con “mellas” de cadena sencilla a ambos lados del Tn, correspondientes cada zona de cadena sencilla a una de las hebras de la corta secuencia diana.
d) El extremo 3' que hay delante de cada mella sirve ahora como cebador para un proceso de síntesis reparativa de ADN. Así pues, a partir de cada “mella” surge una horquilla de replicación, que avanza hacia el interior del transposón, hasta llegar al otro extremo. cuando cada onda replicativa llega hasta el extremo opuesto, se detiene, y vuelve a intervenir una actividad ligasa que sella las cadenas.
e) El resultado final es un cointegrado, en el que los dos replicones originales están unidos a través de dos copias “directas” (en la misma orientación) del transposón.
2)
 Resolución
del cointegrado: la resolvasa específica del
transposón reconoce las dos secuencias res
(una por cada copia del Tn3), y realiza una reacción de recombinación específica
conservativa, análoga a la reacción de escisión del fago l
. El resultado de esta acción es que se regenera el replicón donador, con una
copia del Tn3, y aparece
una nueva copia
del Tn3 en el replicón receptor, limitada por dos copias en la misma orientación
de la secuencia diana. Observar que cada copia del transposón es en
realidad un “mosaico”: debido al fenómeno de resolución, cada cadena de
cada copia consta de una porción de ADN de nueva síntesis, generada en el paso
de formación de cointegrado. Es decir, no existe una copia “vieja” en el
lugar original y una copia “nueva” en el nuevo, sino que las dos copias
tienen partes parentales y de nueva síntesis en cada cadena.
Resolución
del cointegrado: la resolvasa específica del
transposón reconoce las dos secuencias res
(una por cada copia del Tn3), y realiza una reacción de recombinación específica
conservativa, análoga a la reacción de escisión del fago l
. El resultado de esta acción es que se regenera el replicón donador, con una
copia del Tn3, y aparece
una nueva copia
del Tn3 en el replicón receptor, limitada por dos copias en la misma orientación
de la secuencia diana. Observar que cada copia del transposón es en
realidad un “mosaico”: debido al fenómeno de resolución, cada cadena de
cada copia consta de una porción de ADN de nueva síntesis, generada en el paso
de formación de cointegrado. Es decir, no existe una copia “vieja” en el
lugar original y una copia “nueva” en el nuevo, sino que las dos copias
tienen partes parentales y de nueva síntesis en cada cadena.
Muchos transposones, una vez que se han integrado en un replicón, no pueden insertarse de nuevo en ese mismo replicón, sino que han de hacerlo en otra molécula de ADN. Esto se debe a que poseen mecanismos que impiden que el elemento transponible sature de inserciones una misma molécula de ADN. Ejemplos:
|
la proteína codificada por el gen tnpR del Tn3, además de actuar como resolvasa, interviene como represor del gen tnpA (que codifica la resolvasa), y de su propio gen. | |
|
Algunos transposones, como Tn10, sólo se transponen inmediatamente después de que haya pasado por ellos la horquilla de replicación. En esta situación transitoria, el ADN del elemento se encuentra hemimetilado, y al parecer es esta condición la que activa el promotor de su transposasa así como a sus terminales. |
En general, los transposones se pueden considerar como ejemplos de lo que se ha dado en llamar “ADN egoísta”: porciones de material genético cuyo aparente único “objetivo” es perpetuarse en la célula por medio de su capacidad de crear copias transponibles de sí mismo, aunque no estén sometidos a presión selectiva directa por parte del medio ambiente. Sin embargo, si los transposones fueran demasiado egoístas, podrían llegar a “saturar” de sus copias al genomio “hospedador”, llegando a inactivarlo. Parece ser que la evolución ha llegado a una especie de “solución de compromiso” entre la tendencia del transposón de “moverse” ilimitadamente (lo cual provocaría inactivación insercional de numerosos genes), y la necesidad de conservar la integridad de las funciones de la bacteria. Esto se traduce precisamente en la existencia de distintos mecanismos que limitan el número de inserciones de un mismo elemento transponible dentro de un determinado replicón.
Los transposones y secuencias de inserción causan una amplia gama de alteraciones genéticas que pueden a su vez ser fuente de variabilidad genética sobre la que pueden actuar las fuerzas selectivas del ambiente, para “alimentar” los procesos evolutivos.0
Veamos algunas de las alteraciones genéticas propiciadas por elementos genéticos móviles:
1) La más obvia e inmediata es la inactivación insercional de un gen donde se hayan transpuesto: rompen la continuidad del gen, generando mutaciones que inactivan la función de dicho gen. Si el gen forma parte de un operón policistrónico, la mutación tiene efectos polares sobre la transcripción de los genes distales del operón, debido a que el elemento transponible suministra de zonas susceptibles de generar horquillas en el ARNm que actúan como terminadores de transcripción dependientes de r. La inserción de un elemento transponible en un gen u otra zona de material genético se representa indicando el nombre del transposón, seguido de ::, y finalizando con el nombre del gen o de la zona de ADN. Ejemplo, si una copia de IS1 se ha insertado en el gen lacZ, esto se representa como IS1::lacZ.
2) Algunas secuencias IS (p. ej., IS2, IS3), y algunos transposones (como Tn5 y Tn10) en una de sus orientaciones, pueden activar la expresión de genes adyacentes al sitio de integración. Esto se debe a que estos elementos transponibles poseen potentes promotores que propician transcripción “hacia fuera” respecto del propio elemento.
3) Algunos transposones e IS sufren escisiones anómalas que “arrastran” consigo material genético adyacente: esto provoca grandes delecciones en los replicones donde previamente se habían insertado.
4) Los Tn e IS pueden actuar como regiones portátiles de homología:
a) por ejemplo, dos transposones idénticos, situados en lugares diferentes del cromosoma, pueden recombinarse por medio de la enzima RecA de la recombinación general, dando lugar a inversiones del material intercalado (situado entre los dos elementos).
b) Otro caso: dos transposones idénticos, cada uno situado en un replicón distinto, pueden recombinarse por RecA, dando fusión de replicones. Precisamente esta es la base de la producción de cepas Hfr a partir de las cepas F+: como veremos en el tema de conjugación, tanto el plásmido F como el cromosoma poseen copias de determinadas IS; dichas copias son reconocidas con cierta frecuencia por el sistema de recombinación homóloga, que las recombina (recombinación con un solo crossing-over), lo cual lleva a la fusión del plásmido F con el cromosoma bacteriano.
5) Producción evolutiva de transposones complejos a partir de IS o de transposones más sencillos. Imaginen un gen que codifique una resistencia a algún antibiótico; supongamos que un determinado IS se inserta en las cercanías de este gen. Pasado el tiempo, puede ocurrir que otra copia de esa secuencia IS se inserte cerca de ese gen, pero por el otro flanco. Observen que ahora tenemos un transposón compuesto (de los que tienen extremos que son IS). Este nuevo transposón se comporta como una unidad, usando las funciones de transposición de uno de los IS. Esta es precisamente una de las bases genéticas que explican la aparición y diseminación de cepas bacterianas resistentes a antibióticos: desde que el hombre inauguró la era de los quimioterápicos a mediados de este siglo, estamos asistiendo a un auténtico proceso de evolución rápida de transposones complejos, algunos de los cuales portan varios genes de resistencia (frente a varios antibióticos). Los transposones pueden “pasar” a gran variedad de replicones, incluyendo plásmidos conjugativos, que van diseminando al transposón por gran variedad de cepas bacterianas, incluidas patógenas. Como se puede imaginar, esto constituye un problema de primer orden, ya que puede limitar e incluso impedir una adecuada terapia por antibióticos en numerosas infecciones bacterianas. En el tema 18 hablaremos más sobre los plásmidos de resistencia (plásmidos R) y sobre su carácter “modular”, es decir, el hecho de que en buena parte están “cosntruidos” a base de módulos o “cassettes” formados por transposones, IS, transposones anidados (un transposón dentro de otro), etc. Como ya dijimos en el tema 7, aunque los genomas son relativamente estables, la presencia de diversos y variados elementos genéticos transponibles permite un tipo de “flexibilidad” por la que, de vez en cuando se producen nuevas variantes por fusión o agregación de elementos. Es como si los genomas “jugaran al Lego” ensamblando nuevos módulos a partir de otros, y seguramente este es el origen de algunas de las islas genómicas, que como vimos, dotan de ciertas ventajas adaptativas a las poblaciones bacterianas que las poseen (patogenicidad, simbiosis,etc.). En los plásmidos de resistencia a antibióticos es frecuente encontrar casos en los que existe un enorme módulo genético parecido a un gran transposón, que a su vez alberga dentro transposones más sencillos y secuencias de inserción.
En la introducción a este capítulo ya aludimos a los varios destinos que puede experimentar el material genético del exogenote una vez que entra al endogenote:
|
si el exogenote es un replicón funcional (que posee un origen de replicación capaz de ser reconocido por la maquinaria replicativa de la célula receptora), este exogenote permanece como tal replicón en estado autónomo (por ejemplo, plásmidos). | |
|
El exogenote puede recombinarse con el endogote, mediante algunos de los procesos recombinativos descritos en la primera parte de este tema. | |
|
Pero el exogenote puede sufrir otra alternativa: puede ser reconocido como “extraño” por parte de la célula hospedadora, por medio de un fenómeno enzimático llamado restricción, que conduce a la destrucción de este exogenote. Este va a ser el objeto de estudio de esta sección del tema. |
La restricción es un sistema que poseen muchas cepas bacterianas, y representa una especie de “barrera” frente a la permanencia de ADN extraño que hubiera podido entrar por alguno de los sistemas que estudiaremos en los capítulos siguientes. De esta forma, las bacterias evitan la “promiscuidad” en los intercambios genéticos. El posible significado adaptativo de este tipo de sistemas es el de conferir “inmunidad” frente a exogenotes (por ejemplo, fagos) que pudieran poner en peligro la individualidad genética e incluso la superviviencia de la bacteria en cuestión.
La restricción va asociada a un sistema “paralelo” de salvaguardia del ADN de la bacteria frente al propio sistema de restricción. Este sistema de protección del ADN propio se denomina modificación, y mediante él, el ADN queda “marcado” químicamente por metilación de determinadas secuencias.
La restricción y modificación fue descubierta en 1968, a resultas de una serie de estudios sobre la infección del fago l en dos cepas diferentes de Escherichia coli (las llamadas cepas “K12” y “B”):
 Observar que los fagos obtenidos por infección en cada una de las cepas
están “restringidos” a crecer en el mismo tipo de cepa del cual proceden.
Observar que los fagos obtenidos por infección en cada una de las cepas
están “restringidos” a crecer en el mismo tipo de cepa del cual proceden.
Ahora bien, como se puede ver, cuando se intenta infectar una cepa con fagos obtenidos a partir de la otra cepa, la restricción no es total: siempre hay un pequeño porcentaje (del orden de 1 en 10 000) de fagos que “escapan”, y que después de esto pueden crecer en la otra cepa con eficiencia de 100%. Explicación:
|
Cada cepa posee una actividad endonucleasa que reconoce como extraño al ADN del fago crecido en la otra, y lo destruye. A este tipo de endonucleasas se las denomina endonucleasas de restricción, o más abreviadamente, restrictasas. | |
|
Con baja frecuencia, el ADN de un fago puede “escapar” a la restricción de la cepa opuesta a donde ha crecido. Una vez que ha escapado, este ADN es reconocido como “propio” por parte de esta cepa, de modo que este fago podrá crecer con total eficiencia (100%) en dicha cepa. La razón es que el ADN es modificado químicamente por el hospedador, de modo que queda protegido de la correspondiente restrictasa de esa misma cepa. La modificación química consiste en la introducción de grupos metilo (CH3-) en determinadas bases dentro de determinadas secuencias del ADN, lo cual está catalizado por una metilasa específica. |
Se han descrito varios tipos distintos de sistemas de modificación-restricción (M-R), según sus mecanismos de acción generales, pero nosotros sólo trataremos los dos mejor concocidos: M-R tipo I, tipo II.
Fueron los primeros en ser descritos. Precisamente el ejemplo típico lo constituye el caso al que acabamos de hacer referencia en la página anterior. La cepa K12 de E. coli posee el sistema llamado EcoK, y la cepa B posee el llamado EcoB. Lo característico de los sistemas M-R de tipo I es que las actividades de modificacación y las de restricción están producidas por un complejo multiproteico (multimérico), que realiza los dos tipos de actividades. Ejemplo: Complejo de EcoK posee 2 subunidades R (con actividad nucleasa de restricción), 2 subunidades M (con actividad de metilación)y 1 subunidad S (confiere especificidad de reconocimiento de secuencia al complejo).
|
Reconocen secuencias relativamente grandes, que no presentan simetrías. | |
|
La actividad endonucleasa (de las subunidades R) no corta dentro de la secuencia reconocida, sino lejos de ella, a distancias variables (del orden de unos 1000 pb., y en lugares inespecíficos. | |
|
Por otro lado, la actividad nucleasa va acompañada de hidrólisis de ATP | |
|
La actividad metilasa del complejo hace uso de S-adenosil-metionina (SAM) como donador de los grupos metilo. |
Veamos en más detalle el comportamiento del complejo del complejo EcoB. Observen que el complejo se comporta de modo diferente según que actúe sobre ADN de tipo parental previamente metilado en ambos cadenas, o de que actúe sobre ADN hemimetilado (situación que se produce inmediatamente después de la replicación), o sobre ADN no metilado en la secuencia-diana (que sería reconocido como “extraño”):
|
sobre ADN metilado: es reconocido como “propio” (merced a la subunidad S); no sufre ninguna reacción por parte de EcoB. | |
|
sobre ADN hemimetilado: las subunidades M metilan la cadena no metilada (cadena hija de nueva síntesis). | |
|
sobre ADN no metilado: es reconocido como “extraño”. Interviene la actividad restrictasa, con hidrólisis de ATP. El ADN es destruido. |
Observen las diferencias con respecto a los de tipo I:
|
Las actividades metilasa y restrictasa están en proteínas distintas que no forman complejos multifuncionales. | |
|
No requieren ATP. Sólo necesitan iones Mg2+ para funcionar. La metilasa, además, usa SAM como donador de metilos. | |
|
Cada miembro de la pareja de modificación y restricción reconoce la misma secuencia específica de ADN. | |
|
La secuencia reconocida consiste siempre en un corto número de pares de bases. Típicamente consta de 4 o 6 p.b.([2]) que constituyen una secuencia palindrómica o con una simetría patente. | |
|
La metilasa suele ser una proteína monomérica, que introduce grupos metilo en una A o en una G (según la metilasa en cuestión), en ambas cadenas, dentro de la secuencia reconocida. | |
|
La restrictasa suele ser una proteína formada por dos subunidades del mismo tipo, ensambladas simétricamente. Muchas restrictasas cortan dentro de la secuencia, dejando extremos protuberantes mutuamente cohesivos, o extremos romos (según la restrictasa en cuestión). Algunas restrictasas dejan extremos protuberantes 5', mientras que otras dejan extremos protuberantes 3'. |
|
|
El sistema de restricción-modificación EcoRI: tanto la restrictasa como la metilasa de este sistema reconocen la misma secuencia palindrómica. En a) se muestra dónde corta la restrictasa en cada una de las cadenas. En este caso se generan extremos protuberantes en 5’ (de cuatro bases). En b) se muestran las dos adeninas (una por cadena) que son metiladas dentro de la secuencia reconocida por la enzima modificante. |
El mecanismo general de las restrictasas de tipo II es más sencillo que las de tipo I: cada subunidad del dímero reconoce la misma secuencia, presente en cada una de las partes especulares del palíndromo, y realiza un corte en un lugar específico, que obviamente tiene su correspondiente en la otra cadena de la otra mitad del palíndromo.
Existen cientos de restrictasas de tipo II (y se están descubriendo continuamente). Más de la tercera parte de las cepas bacterianas presentan al menos un sistema M-R de tipo II. Muchas de ellas son codificadas a partir de genes situados en plásmidos.
Se habla de isosquizómeros para referirse a aquellas restrictasas que, siendo diferentes y procediendo de especies distintas, reconocen y cortan la misma secuencia, dejando el mismo tipo de extremos.
Las restrictasas de los sistemas de tipo II se nombran de modo mnemotécnico a partir de la cepa de origen, usando las iniciales de la especie de la que derivan. Por ejemplo, la llamada EcoRI procede de cierta cepa de Escherichia coli. Si una cepa posee más de un sistema, a las iniciales se añaden números romanos. Ejemplo, de Haemophilus influenzae tenemos los sistemas HindII y HindIII.
No hace falta insistir en la importancia práctica de las restrictas de tipo II, como herramientas básicas -junto con la ADN ligasa- en la Ingeniería Genética. El biólogo cuenta con un impresionante “arsenal” -comercializado- de enzimas (no sólo las que acabamos de citar) para manipular a placer el ADN de cualquier ser vivo...(pero esta es ya otra historia, que el alumno interesado irá conociendo en esta y en otras asignaturas).
La transformación se puede definir como la variación hereditaria de una célula bacteriana susceptible, originada por la captación de ADN desnudo libre en el medio, con la posterior recombinación del exogenote con el genomio de la célula en cuestión (endogenote). Tras la transformación, la célula que ha recibido el ADN se suele denominar transformante.
Recordemos brevemente los hitos históricos que jalonan el descubrimiento de la transformación en bacterias:
|
La transformación fue el primer proceso de transferencia genética que se describió en los procariotas, y este hallazgo constituyó la base para suministrar el primer dato incontrovertible acerca de la naturaleza química del material genético: a partir de 1944 empezó a quedar claro que era el ADN el portador de la información genética de los seres vivos. | |||||||||
|
Todo comenzó con los trabajos de Griffith (1928) sobre el neumococo. Recuérdense sus clásicos experimentos con las cepas S (lisas, capsuladas y virulentas) y R (rugosas, no capsuladas y avirulentas), inoculadas en ratones:
| |||||||||
|
Ni Griffith ni los investigadores de la época se dieron cuenta de la importancia de este resultado, ni sacaron la conclusión pertinente: que una sustancia que confiere una nueva propiedad heredable a un organismo (la “sustancia transformante” procedente de las S muertas y que captan las R vivas) debe a su vez ser capaz de replicación. (De hecho, y aunque ahora nos parezca mentira, Griffith pensaba que el principio transformante era el polisacárido capsular de las cepas S, ausente en las cepas R). | |||||||||
|
En 1944 el equipo formado por Avery, MacLeod y McCarty descubrió la naturaleza del misterioso “principio transformante”: el ácido desoxirribonucleico (ADN). Ellos lograron desarrollar un sistema in vitro, en el que iban ensayando diversas fracciones (con diferentes componentes) procedentes de las células S virulentas frente a células R vivas. (A pesar de los elegante experimentos, la comunidad científica no los aceptó inmediatamente ([3]), en parte debido a que se suponía más lógico que el material de la herencia fuera algún tipo de proteínas. Al fin y al cabo, las proteínas tienen una enorme variedad, a base de 20 tipos de aminoácidos. ¿Qué diablos podía significar que el ADN fuera el material genético, cuando se sabía que era una molécula monótona e insulsa a base de sólo cuatro tipos de bases nitrogenadas en proporciones casi semejantes?). | |||||||||
|
El siguiente paso importante sobre la transformación lo da Hotchiss (1949), que realiza ensayos in vitro al estilo de los de Avery y compañía, pero con ADN extremadamente puro. Los remisos contestatarios no tuvieron más remedio que empezar a batirse en retirada. Como sabemos, los últimos incrédulos (que en los años 40 eran la mayoría) sólo se convirtieron (tras el correspondiente arrepentimiento y propósito de enmienda) cuando Hershey y Chase (1952) realizaron sus ingeniosos experimentos de marcado del ADN y de las proteínas de un fago. Poco más tarde irrumpieron Watson y Crick con su modelo de la doble hélice, que sugería el modo de duplicación de la información genética ...pero en fin, todo esto nos aleja de nuestra historia, y no queremos convertir nuestra narración en un caso de “novela dentro de otra novela”, al estilo de Cervantes. |
1. El ADN, para que sea capaz de transformar, ha de ser de cadena doble.
2. Para cada evento de transformación basta la interacción de una sola molécula de ADN con la célula receptora. Por otro lado, el número de moléculas de ADN que puede tomar una misma célula es limitado, y presenta una cinética de saturación (meseta que indica que ya no se pueden captar más moléculas). Observen la cinética de orden 1 en la primera parte de la curva. A partir de cierta concentración de ADN transformante, se da la meseta de saturación (no aumenta el nº de colonias transformadas).
3. No todas las células de un mismo cultivo son transformables en cualquier momento del ciclo de crecimiento. La competencia es el estado fisiológico característico que permite captar ADN del medio exterior. Este estado de competencia es diferente para cada especie bacteriana capaz de experimentar transformación, y dentro de cada especie está influido por una serie de parámetros como:
|
densidad celular del cultivo | |
|
temperatura | |
|
pH | |
|
nutrientes (fuentes de C o de N, iones...) |
Ejemplos:
|
En Streptococcus pneumoniae la competencia afecta al 100% de las células en fase exponencial. | |
|
En cambio, en Bacillus subtilis solamente una minoría (1-20%) de células se hacen competentes, y sólo durante la fase estacionaria. |
4. Se denomina fase de eclipse durante la transformación al período transitorio durante el cual, si se extrae el ADN recién entrado (exogenote), este ADN es incapaz de volver a transformar. Es decir, tras su entrada a la célula receptora, el ADN del exogenote parece haber “desaparecido” transitoriamente a efectos de capacidad transformadora (ya veremos por qué).
En la transformación natural de diversas especies bacterianas se dan una serie de fases comunes a todas ellas, aunque en cada caso existen rasgos propios:
1. Competencia
2. Unión y entrada del ADN exógeno a la célula
3. Fase de eclipse
4. Destino del ADN exógeno (exogenote): recombinación con el endogenote.
En la naturaleza, la fuente de ADN exógeno suele ser la lisis espontánea de células de la misma especie, que actúan como “donadoras” (una especie de inmolación o suicidio altruista, para mayor gloria -o sea, para mayor superviviencia- de la especie). En otros casos no hay lisis, sino que parte de la población bacteriana extruye ADN, que pasa al medio.
Existen varios géneros con especies que poseen sistemas naturales de transformación:
|
Entre las bacterias Gram positivas los ejemplos más estudiados son los de:
| |||||||||||||||
|
Entre las bacterias Gram negativas:
|
A continuación describiremos algunos de los sistemas de transformación mejor conocidos. Primero abordaremos los de bacterias Gram positivas, y posteriormente aludiremos más brevemente a los de Gram negativas, comentando las diferencias de éstas respecto de las primeras.
Los sistemas de estas bacterias Gram positivas son inespecíficos respecto de la captación de ADN exógeno: no distinguen entre ADN homólogo (de la misma especie) o ADN heterólogo (de otras especies). Sin embargo, aunque físicamente puede entrar ADN heterólogo, éste se recombina posteriormente con el endogenote sólo en el caso de presentar con él un grado de homología suficientemente alto. Únicamente se recombinan moléculas de ADN de la misma especie (o en algunos casos, de especies muy cercanas).
Veamos comparativamente los procesos de transformación en el neumococo (S. pneumoniae) y en B. subtilis:
Como dijimos, el estado de competencia se encuentra condicionado por una serie de factores, como densidad celular, temperatura, pH, etc.
En esta bacteria la competencia surge de repente al final del periodo de crecimiento exponencial (fase logarítmica), afectando prácticamente a la totalidad de las células del cultivo (100% de células competentes), se mantiene durante solo 10-15 min y luego decae tan rápidamente como había surgido.
|
Conforme las células van creciendo (se van multiplicando), van sintetizando y excretando al medio un péptido específico de 17 aminoácidos llamado CSP (iniciales de péptido estimulador de la competencia)
| |||||||
|
Al llegar a cierta concentración extracelular, el CSP se une a un histidín-proteín-quinasa sensora de membrana (ComD), que tras autofosforilarse fosforila al regulador de respuesta (ComE). | |||||||
|
La proteína ComE~P activa la transcripción de varios operones:
| |||||||
|
La ARN-polimerasa provista de ComX transcribe una serie de operones tardíos de competencia, que codifican la maquinaria para la captación del ADN exógeno y su entrada y recombinación.
|
La fase de eclipse se pone de manifiesto en el laboratorio por el hecho de que el ADN exógeno se convierte a una forma resistente a la DNasa. Esto significa que este ADN ha sido transportado desde el medio externo al interior celular (o al menos a algún “compartimiento” donde se ve protegido del ataque de la nucleasa).
|
Hay una actividad endonucleasa (EndA) específica de la competencia, que introduce primero cortes en una hebra y luego en ambas. Esto suministra extremos libres de ADN. Una de las cadenas entra al citoplasma, probablemente unida por su extremo 3’ con una proteína. La otra cadena es degradada hasta oligonucleótidos. | |||||
|
Conforme va entrando, la cadena (de unas 6.000 bases por término medio) va siendo recubierta por monómeros (de unos 20 kDa) de una proteína específica, hasta formarse el complejo de eclipse (entre ese ADN de c.s. y las proteínas). Dicho complejo cumple dos funciones:
|
(Si utilizáramos este ADN de c.s. del complejo de eclipse para intentar una nueva transformación, no podría ser reconocido por el complejo receptor que describimos anteriormente. Esta es la razón de la existencia de lo que hemos denominado período de eclipse).
En cada célula receptora pueden entrar al mismo tiempo varios fragmentos de exogenote, que conjuntamente pueden representar hasta el 5% del genoma.
Se da una recombinación homóloga (dependiente de RecA, que previamente había sido inducida por la competencia) de la cadena sencilla del exogenote con la zona complementaria del endogenote, previo desplazamiento y ulterior degradación de la cadena homóloga de éste. Esta recombinación homóloga es muy eficiente (un 50% del ADN transportado se integra, y puede afectar hasta al 10% del genoma receptor). Como resultado de esta integración se forma un heterodúplex. Si el heterodúplex posee malos emparejamientos de bases (debido a que la secuencia del exogenote no es idéntica a la del endogenote), actúa un sistema de reparación de malos emparejamientos, denominado sistema Hex. Los resultados de ese sistema son diferentes, según que actúe antes o después de que pase la onda de replicación:
|
Si el sistema Hex actúa de modo pre-replicativo, escinde toda la cadena del exogenote y sintetiza ADN usando como molde la cadena del endogenote. De este modo se restablece el ADN original del endogenote, | |
|
Pero si antes de que intervenga el sistema Hex pasa la onda de transcripción por el heterodúplex, cada cadena servirá de molde para una nueva doble hélice, pasando cada una a una célula hija. De este modo, a partir de este evento de recombinación surge una progenie mixta: existe un subclon recombinante y otro que no lo es. | |
|
Es de destacar que, el sistema Hex que restablece el mensaje original es más eficiente cerca de ciertos sitios del ADN (sitios hex, reconocidos por cierta proteína del sistema). Si el exogenote se integra lejos de uno de los sitios reconocidos por Hex, tiene más probabilidades de escapar a la corrección, y por lo tanto toda la progenie podrá heredar el marcador nuevo procedente del exogenote. | |
|
Puesto que cada célula, como dijimos antes, puede captar simultáneamente varios fragmentos de exogenote, su eventual recombinación y escape al sistema Hex significa que una bacteria transformada puede adquirir uno o varios alelos nuevos al mismo tiempo. |
Las principales diferencias respecto del modelo del neumococo, en esta fase inicial de competencia, son las siguientes:
|
La competencia se desarrolla después de la fase exponencial, durante el inicio de la fase estacionaria. | |||||||
|
Los “cultivos competentes” son heterogéneos: existe una minoría de células (< 25%) auténticamente competentes, y se encuentran en un estado metabólico alterado:
|
La competencia en B. subtilis está sometida a tres tipos de controles:
1. Primer control, según la fase de crecimiento por un sistema de percepción de quórum
2. Segundo control, de tipo nutricional. Para que se induzca la competencia, el medio de crecimiento ha de poseer glucosa como fuente de C, junto con una mezcla de aminoácidos. Las señales nutricionales que detecta la bacteria para provocar el estado de competencia parecen depender (al menos en parte) del agotamiento de algunos aminoácidos y de alguna señal derivada del metabolismo de la glucosa.
3. Tercer control, según el tipo celular. Como acabamos de decir, existen células competentes y no competentes. Es aún un misterio cómo se desarrolla este “dimorfismo” para la competencia. Lo que sí se sabe es que durante todo el crecimiento, algunas células (probablemente las no-competentes) excretan al medio ADN de alto peso molecular, que será la fuente del ADN transformante.
De esos controles, el mejor conocido hasta ahora es el relacionado con la densidad celular, basado en la detección de feromonas peptídicas, y que depende de un sistema de quórum-sensing y de un sistema regulador de dos componentes
La competencia se desarrolla en la transición entre la fase exponencial y la de esporulación, y se inicia por dos péptidos extracelulares: ComX y CSF (iniciales de factor de competencia y esporulación), que durante la fase exponencial van siendo liberados al medio e incrementan su concentración en paralelo con la densidad celular.
Sistema de ComX:
|
Mientras B. subtilis va creciendo, va segregando un péptido, denominado ComX. Obviamente, en un sistema cerrado, dicho péptido se va acumulando en el medio. | |
|
Cuando se alcanza cierto nivel de ComX (y esto sólo sucede a partir de ciertas densidades altas de cultivo), interacciona con una proteína de membrana denominada ComP. ComP es una histidín-proteín-quinasa, del tipo de los sensores autofosforilables. Cuando se le une CompX, la ComP se autofosforila, y entonces fosforila a su vez a una proteína citoplásmica llamada ComA. | |
|
La ComA~P es un regulador de respuesta, que en ese estado fosforilado actúa como activador de la transcripción de varios genes, incluyendo el gen comS. | |
|
La proteína ComS se une a un aparato proteolítico, causando la liberación de ComK, y protegiendo a esta última de la proteolisis. | |
|
A su vez, el ComK liberado actúa como activador de la transcripción de su propio gen, de modo que en poco tiempo aumenta su concentración, y entonces el ComK activa la transcripción de una serie de genes de competencia tardíos, que codifican las proteínas necesarias para establecer ese estado de competencia |
Sistema de CSF:
Mientras ocurre lo anterior, en paralelo se da otra serie de fenómenos: el péptido CSF, por su parte, al llegar a cierta densidad, interacciona con un transportador de membrana especializado en oligopéptidos (oligopéptido-permeasa), con lo cual el CSF entra al citoplasma. Una vez allí, el CSF inhibe la fosfatasa que quita el fosfato de la ComA. Por lo tanto, este sistema confluye con el anterior, manteniendo niveles altos de ComA~P (el regulador de respuesta).
(Curiosamente, parecen existir vínculos entre desarrollo de la competencia y esporulación y no sólo mera coincidencia de sus respectivos desencadenamientos en el tiempo, ya que hay genes que intervienen en la regulación de ambos procesos).
|
El ADN exógeno interacciona en primer lugar, a nivel de pared celular, con un sistema de pseudopelos formado por proteínas ComG: una NTPasa de tráfico (ComGA), una proteína de membrana politópica (ComGB) y pseudopilinas. Es posible que este sistema modifique de algún modo la pared celular, quizá creando un canal, por el que el ADN puede llegar hasta el lado externo de la membrana citoplásmica. | |||||||
|
Entonces, el ADN de cadena doble llega hasta la maquinaria de translocación de ADN a nivel de membrana:
| |||||||
|
Como acabamos de ver, aunque la maquinaria de translocación de ADN une ADN de cadena doble, al citoplasma solo entra cadena sencilla. Probablemente este sistema va acoplado a una degradación de ADN. |
4,2.3.1 TRANSFORMACIÓN EN HAEMOPHILUS Y NEISSERIA
Una diferencia característica de los sistemas de estas bacterias Gram-negativas con respecto a los de Gram-positivas es que el estado de competencia no depende de señal activadora segregada al medio, sino que se induce internamente.
Otra importante diferencia es que sistema de entrada del ADN discrimina entre ADN de la especie (o, en todo caso de especies cercanas) y ADN de otras procedencias: solamente capta ADN de especies del mismo género, y con gran eficiencia. La secuencia se denomina DUS (DNA uptake sequence), y está repetida varias veces a lo largo del genoma.
|
Para Haemophilus la DUS es 5'AAGTGCGGTCA-3'. Dicha secuencia aparece repetida y repartida en el genoma de H. influenzae unas 600 veces, a una media de una copia cada 4 kb. Como se puede comprobar, esta frecuencia es muy superior (3 órdenes de magnitud) a la que predice el cálculo de probabilidades en el caso de que apareciera aleatoriamente (4000 kb). Así pues, el genoma de estas bacterias está “preparado” (mediante la existencia sobreabundante y no-aleatoria de esta secuencia) para participar en eventos de reconocimiento específico por parte de receptores encaminados a la entrada de ADN propio por medio de fenómenos de transformación. | |
|
Para Neisseria: 5’-GCCGTCTGAA-3’. Ahora que se ha secuenciado el genoma (2.15 Mb) de una cepa de N. gonorheae se sabe que contiene 1965 copias de DUS, lo que supone una media de una DUS cada 1096 pb. |
La competencia afecta al 100% de la población. En Haemophilus se da en la fase estacionaria, mientras que en las especies del género Neisseria las células se mantienen competentes durante todo el ciclo de crecimiento, cosa excepcional dentro de los sistemas naturales de transformación.
Veamos un resumen de lo que se sabe de la transformación del gonococo (N. gonorhoeae).
¿De dónde procede el exogenote? En N. gonorheae intervienen dos mecanismos: un sistema de secreción de tipo IV y la autolisis.
Hoy se sabe que el 80% de los aislados de gonococo poseen islas genómicas que codifican sistemas de secreción de tipo IV (T4SS), que como ya vimos en otro capítulo, muestran trazas de haber sido adquiridas por transferencia horizontal. Estos sistemas parece que son responsables de una parte del ADN exógeno que luego será captado por bacterias competentes que comparten el mismo medio.
El restante 20% de cepas de gonococo, y todas las de meningococo liberan ADN solamente por autolisis. Se han estudiado algunas muramidasas en estas especies, pero no está claro su papel en esta autolisis. Incluso existen ciertos indicios de que pueda haber un mecanismo de “muerte celular programada”, que afectaría a una fracción aleatoria del cultivo.
Aunque como decíamos, estas especies solo son transformadas por ADN de la misma especie o especies relacionadas a través del reconocimiento de las secuencias DUS repartidas por el genoma, no se conoce el supuesto receptor específico.
Se sabe que en la captación del ADN está implicado un sistema de “pseudopelos” relacionado con los pelos de tipo IV (T4P) y los sistemas de secreción de tipo II. Al parecer, la captación no requiere pelos funcionales, pero sí parte de las proteínas implicadas en la formación de esos pelos. Se ha especulado con que el supuesto receptor para las DUS (que debería estar en la membrana externa) al unirse al ADN de forma específica permite la apertura de la secretina PilQ de la membrana externa, que forma parte del “pseudopelo”. La función de este pseudopelo sería conducir el ADN hasta la maquinaria de translocación a nivel de membrana citoplásmica, que permitirá que el exogenote entre en el citoplasma.
Descripción del hipotético sistema de entrada basado en pseudopelos de tipo IV: Se trataría de un pseudopelo de competencia, relacionado con el pelo de tipo IV, pero no idéntico. (No se necesitan pelos funcionales para la transformación, pero sí parte de su maquinaria).
El extremo distal consistiría en la secretina PilQ de membrana externa. La secretina se ensambla en la membrana externa formando multímeros con forma de donut, que dejan un canal interior. En el caso de un pelo de tipo IV, esa canal permite que el pelo pueda proyectase desde el periplasma hasta el exterior (el pelo atraviesa el canal de la secretina). En el caso de la transformación, parece que la secretina igualmente alberga un pseudopelo cuyo componente mayoritario es la pilina PilE (como en el pelo de tipo IV), y probablemente va acompañado de la proteína estabilizante PilC.
El pseudopelo se ancla a la membrana citoplásmica probablemente a través de la proteína politópica PilG, que a su vez tiene asociado, por el lado citoplásmico de la membrana la NTPasa de tráfico PilF.
El modelo (Chen y Dubnau, 2004):
|
las secuencias DUS del ADN exógeno son reconocidas por un receptor específico (sin identificar aún), y pudieran inducir la apertura del canal de la secretina; | |
|
El ADN pasa al pseudopelo de competencia, interaccionando con la secretina PilQ de la membrana externa. El ADN quizá entre por el canal del polímero en donut de la secretina, una vez abierto. | |
|
De esta forma el ADN entraría al periplasma, y probablemente estaría interaccionando con el pseudopelo de pilina PilE, que está inserto en la membrana citoplásmica mediante la PilG, asociada a su vez con la NTPasa PilF. | |
|
Todo ello facilitaría la interaccion del ADN con la translocasa de membrana. |
La maquinaria de translocación a través de la membrana
Se han identificado homólogos de lo visto en B. subtilis:
|
ComE es el ortólogo de ComEA. En el genoma de H. gonorhoeae existen 4 copias del gen comE, cuya sucesiva mutación va disminuyendo la eficiencia de la transformación. Se ha propuesto que se une al ADN en el periplasma, sirviendo de “puente” entre el pseudopelo y la maquinaria a nivel de membrana. | |
|
ComA es el ortólogo de ComEC. Es una proteína politópica de membrana, que quizá forma un canal para el transporte del ADN al citoplasma. | |
|
(No se ha identificado en Gram-negativas ortólogos de la ATPasa ComFA de B. subtilis). |
La situación sobre el procesamiento del exogenote al pasar por la membrana no está tan clara como en Gram-positivas. Parece que parte del ADN cromosómicos entra como cadena sencilla (y no doble, como se pensaba), pero no se han identificado aún las actividades enzimáticas de esa conversión. Ese ADN de cadena sencilla parece que se genera ya en el periplasma. Al entrar de esta manera, queda protegido frente al arsenal de endonucleasas (que en cambio, sí afectan al ADN plasmídico, que por lo tanto sí pudiera entrar como ADN de cadena doble). El tamaño del ADN importado al citoplasma parece estar en torno a 10 kb.
En el gonococo parecen darse las dos ramas de la recombinación homóloga dependientes de RecA (rama RecBCD y rama RecF). Los gonococos recA- no son transformables, y la recombinación parece depender de la rama RecBCD, pero no de la RecF.
La transformación de este patógeno gástrico humano es diferente a las vistas hasta ahora. Implica un aparato relacionado con los sistemas de secreción de tipo IV. (Estos sistemas están implicados en el sistema Vir de transferencia conjugativa del ADN-T entre Agrobacterium y la planta y en la exportación de proteínas al medio o al hospedador eucariótico por parte de ciertos patógenos Gram-negativos).
En H. pilori existe un operón comB, con cuatro ORF homólogas de vir de A. tumefaciens. ComB7 es una lipoproteína quizá de la membrana externa, y forma un puente disulfuro con ComB9. ComB8-ComB10 están estrechamente asociadas con la membrana citoplásmica y probablemente constituyan un puente entre ésta y la membrana externa. La ATPasa es ComB4. La translocación del ADN al citoplasma parece depender de un ortólogo de ComEC de B. subtilis, es decir, H. pilory usa un sistema especial para captar el ADN, pero lo transloca al citoplasma mediante mediante un sistema similar al de las otras bacterias competentes.
Como acabamos de ver, los sistemas naturales de transformación representan mecanismos bastante sofisticados de transferencia génica en ciertas bacterias, y no deben en absoluto considerarse como meras “curiosidades” o “accidentes” de algunos procariotas. Géneros como Haemophilus, Streptococcus o Bacillus presentan exquisitos refinamientos moleculares encaminados, en última instancia, a facilitar expresamente la recombinación genética tras un proceso específico de transformación, induciendo una serie de funciones totalmente exclusivas para tal propósito. Estos mecanismos han debido de evolucionar debido probablemente a ciertas presiones selectivas del ambiente, y han sido “aprovechados” para producir un plus de diversidad genética, que debe de jugar un papel importante en la dinámica ecológica de las especies que lo poseen.
|
Así, por ejemplo, parece ser que el sistema de transformación de Neisseria gonorrhoeae (el gonococo) permite que esta bacteria cambie más fácilmente determinadas moléculas de superficie, de modo que estos cambios antigénicos suponen una ventaja adaptativa para evadir el sistema inmune del hospedador mamífero. El genoma de una cepa secuenciada del gonoco muestra seis islas genómicas, probablemente adquiridas por transformación. | |
|
Lo mismo se puede decir del neumococo, en el que la transformación parece tener un gran papel para dotarlo de flexibilidad adaptativa: Por ejemplo, le permite producir distintos y cambiantes tipos de cápsulas, que le ayudan en la invasión de su hospedador humano, y disponer de genes “mosaico” para proteínas de unión a penicilina, PBPs, con menor afinidad hacia el antibiótico, lo que les permite resistirlo. Además, puesto que S. pneumoniae convive con otras especies del género Streptococcus, y puesto que su sistema de transformación deja entrar cualquier tipo de ADN, en esta especie es la recombinación homóloga (que se permite con ADN cercano evolutivamente) la encargada de “discriminar”, pero permitiendo experimentos naturales en los que se recombinan exogenotes de especies cercanas. Hoy existen pruebas de que el neumococo evoluciona y se adapta en sus mecanismos de virulencia en parte gracias a la transformación interespecífica (con ADN de otras especies de estreptococos, como S. oralis, S. mitis). De hecho, parece que el sistema del neumococo está “pensado” para maximizar la transformación con ADN heterólogo: en la cepa silvestre Hex+ de neumococo, la eficiencia de transformación es proporcional a la divergencia entre exo y endogenote. |
Una pregunta más difícil de responder es la del origen evolutivo de la capacidad de transformación. Aquí caben diversas hipótesis.
|
Hipótesis de la reparación de ADN: sugiere que la capacidad de competencia apareció en B. subtilis. (esporulante) como un sistema de obtener moldes de ADN para la reparación. La argumentación sigue el siguiente curso: se sabe que la competencia en esta especie está regulada por controles que son en parte comunes con los del desencadenamiento de la esporulación (recordar que decíamos que en B. subtilis la competencia se induce en fase estacionaria, lo cual también ocurre con la esporulación). Por otro lado, también se recordará que al final de la fase exponencial ocurren grandes cambios metabólicos en esta bacteria, siendo uno de ellos el que pasa de un metabolismo fermentativo a uno oxidativo. En el metabolismo oxidativo se producen numerosos radicales libres, que pueden provocar daños en el ADN. Por lo tanto -concluye esta hipótesis- , no sería extraño que la competencia hubiera evolucionado como una forma de obtener ADN de repuesto, que capatarían ciertos individuos de la población, para usarlos en la reparación de sus genomios. Posteriormente el sistema se refinaría y seviría para incrementar la variabilidad genética y la capacidad de adaptación. (De hecho, en B. subtilis y en S. pneumonie se ha visto que el gen recA de la recombinación homóloga se induce en respuesta al desarrollo de la competencia. | |
|
Hipótesis de la nutrición: Quizá pudo surgir como una manera de obtener nutrientes adicionales a partir de ADN exógeno. | |
|
Hipótesis de la recombinación: pudo haber una presión selectiva que favoreciera el desarrollo de un mecanismo de intercambio y recombinación de ADN, por los que las poblaciones de ciertas bacterias podrían poner a prueba nuevas combinaciones de genes (algo parecido a lo que pasó con el desarrollo de la sexualidad en los eucariotas). Ello aumentaría la diversidad y podría acelerar la evolución. De hecho, algunas de las especies transformables de modo natural “excretan” ADN durante el crecimiento. Ese ADN, captado en el caso de Haemophilus o Neisseria de un modo muy específico, puede favorecer la adaptación de otros miembros de la especie. De hecho, en esas y otras especies (incluyendo el neumococo), la transformación aumenta la diversidad antigénica, y por lo tanto son mejores las probabilidades de sobrevivir en el huésped al que parasitan. Puesto que en la recombinación solo unas cuantas combinaciones van a ser beneficiosas, se necesitan altas densidades celulares para aumentar la probabilidad de que aparezcan y se seleccionen esas raras combinaciones. Ello justificaría el que en al menos algunas especies la competencia dependa de un mecanismo de percepción de quórum. |
Se define la transfección como la infección de una célula hospedadora mediante ADN obtenido de un virus. El ADN del virus así introducido, al expresar su programa genético en la célula, termina desencadenando la producción de nuevas partículas de virus dentro, que al igual que en la infección “clásica” por virus completos, conduce a la muerte y lisis de la célula, con la liberación de la progenie de nuevos viriones. Las características de unión de ADN y entrada dependerán del sistema de células competentes que se esté empleando, mientras que el resultado de la transfección depende (al igual que en los ciclos líticos naturales de los virus) del programa del virus en cuestión.
Muchas especies bacterianas carecen de sistemas naturales de transformación. En los últimos años se ha logrado desarrollar sistemas artificiales para producir “células competentes” en algunas de ellas (como Escherichia coli, Salmonella typhimurium y algunas especies de Pseudomonas), lo cual ha hecho avanzar los estudios genéticos, especialmente mediante manipulaciones in vitro: por ejemplo, la transformación artificial de E. coli con ADN plasmídico es una de los métodos básicos de la Ingeniería Genética.
La obtención de células competentes en E. coli consiste en ir concentrando un cultivo recogido en fase logarítmica, mediante lavados sucesivos en una solución fría (4ºC) de Cl2Ca, tras lo cual la mezcla de células y ADN se someten a unos 2 min. a 42ºC (choque por calor). Este tratamiento altera las envueltas (sobre todo la membrana externa) y aumenta su permeabilidad al ADN. Los plásmidos entran a la célula como moléculas de cadena doble intactas, mientras que los ADN lineales, que entran de la misma forma, son degradados por nucleasas citoplásmicas (RecBCD). Los transformantes se suelen seleccionar en base a la resistencia a algún o algunos antibióticos conferidos por los plásmidos artificiales usados en Ingeniería Genética.
En otras bacterias (p. ej., en ciertas Gram-positivas) el método consiste en obtener protoplastos en medio hipertónico, y posterior tratamiento de éstos mezclados con el ADN con un agente como el polietilénglicol, que fomenta la fusión de protoplastos y el englobamiento de ADN exógeno.
Más recientemente se ha introducido la técnica de la electroporación: las células se someten a cortos pulsos de corriente eléctrica de alto voltaje, lo cual altera las envueltas y permite la captación de ADN en una amplia variedad de bacterias, tanto Gram-positivas como Gram-negativas.
![]()
![]()
[1].- Esta secuencia se encuentra aleatoriamente distribuida por el genomio de E. coli de modo que aparece una vez cada 5000-10000 pares de bases.
[2].- Existen sistemas de tipo II que reconocen secuencias más largas, de 8 o 9 pares de bases. Lógicamente, por puro cálculo de probabilidades, estas secuencias aparecen aleatoriamente con menor frecuencia en los genomios. A las restrictasas que reconocen secuencias mayores de 6 p.b. se las denomina "cortadoras raras".
![]()